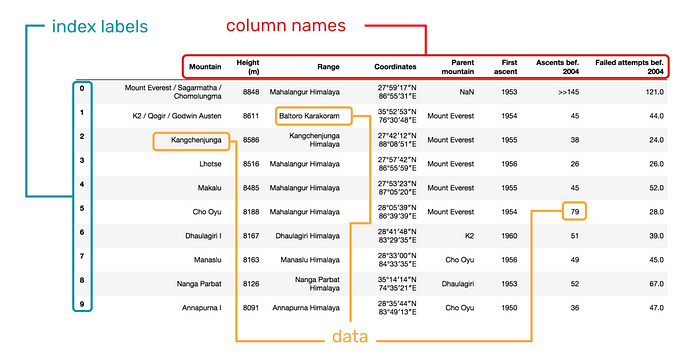
Introduction aux structures de données pandas : les DataFrames (tableaux bidimensionnels)
Commençons par importer la bibliothèque Pandas (et la bibliotèque NumPy) avec les alias classiques pd et np :
La deuxième structure fondamentale de Pandas est le DataFrame. Elle peut être considérée comme une généralisation d’une matrice NumPy, où les lignes et les colonnes sont identifiées par des étiquettes, ou encore comme un dictionnaire de Series partageant le même index.
Contrairement à une matrice, les colonnes peuvent être de types différents.
Un DataFrame est composé des éléments suivants :
- l’indice de la ligne ;
- le nom de la colonne ;
- la valeur de la donnée ;
1 Création d’un DataFrame
On peut créer un DataFrame
à partir d’une matrice NumPy (en précisant éventuellement les noms des colonnes et des lignes)
à partir d’un fichier CSV, Excel, SQL, etc.
colonne par colonne à partir
- d’une seule Series
- d’un dictionnaire de Series ou d’un dictionnaire de listes (chaque élément du dictionnaire est une colonne)
ligne par ligne à partir
- d’une liste de Series ou d’une liste de dictionnaires (chaque élément de la liste est une ligne)
1.1 À partir d’un tableau NumPy à deux dimensions
On peut aussi créer un DataFrame à partir d’un tableau NumPy à deux dimensions.
ARRAY = np.array([ [100, 50, 20, 100, 50],
[1000, 300, 400, 50, 200],
[10, 20, 5, 200, 25] ])
df3 = pd.DataFrame( data=ARRAY )
df3| 0 | 1 | 2 | 3 | 4 | |
|---|---|---|---|---|---|
| 0 | 100 | 50 | 20 | 100 | 50 |
| 1 | 1000 | 300 | 400 | 50 | 200 |
| 2 | 10 | 20 | 5 | 200 | 25 |
On peut spécifier les noms des colonnes et des lignes :
ARRAY = np.array([ [100, 50, 20, 100, 50],
[1000, 300, 400, 50, 200],
[10, 20, 5, 200, 25] ])
columns = ["Facebook", "Twitter", "Instagram", "Linkedin", "Snapchat"]
indices = ["Budget", "Audience", "CPM"]
df3 = pd.DataFrame( data=ARRAY,
columns=columns,
index=indices
)
df3| Snapchat | |||||
|---|---|---|---|---|---|
| Budget | 100 | 50 | 20 | 100 | 50 |
| Audience | 1000 | 300 | 400 | 50 | 200 |
| CPM | 10 | 20 | 5 | 200 | 25 |
1.2 Colonne par colonne à partir …
1.2.1 … d’une seule Series
population = pd.Series( data=[8.516, 67.06, 328.2, 1_386],
index=["Suisse", "France", "USA", "Chine"] # les index seront les labels des lignes
)
display( population )
# df0 = pd.DataFrame( data=population )
df0 = pd.DataFrame( data=population,
columns=["Population"]
)
display(df0)Suisse 8.516
France 67.060
USA 328.200
Chine 1386.000
dtype: float64| Population | |
|---|---|
| Suisse | 8.516 |
| France | 67.060 |
| USA | 328.200 |
| Chine | 1386.000 |
display(df0.values) # un array 2D numpy
display(df0.index) # un objet de type Index
display(df0.columns) # un objet de type Index
display(df0["Population"]) # une Series
display(df0["Population"]["Suisse"]) # DataFrame["colonne"]["ligne"]array([[ 8.516],
[ 67.06 ],
[ 328.2 ],
[1386. ]])Index(['Suisse', 'France', 'USA', 'Chine'], dtype='object')Index(['Population'], dtype='object')Suisse 8.516
France 67.060
USA 328.200
Chine 1386.000
Name: Population, dtype: float648.516Si les éléments index et columns d’un DataFrame ont leurs attributs name définis, ceux-ci seront également affichés :
| Caractéristiques | Population |
|---|---|
| États | |
| Suisse | 8.516 |
| France | 67.060 |
| USA | 328.200 |
| Chine | 1386.000 |
1.2.2 … à partir d’un dictionnaire de Series {label_1: serie_1, label_2: serie_2, etc} ou de listes de valeurs {label_1: [val_1, val_2, etc], label_2: [val_1, val_2, etc], etc}
Chaque Series devient une colonne du DataFrame.
Les clés du dictionnaire sont les noms des colonnes et les valeurs associées sont les Series ou listes de valeurs pour chaque colonne.
Les index peuvent être passés ou générés automatiquement.
Le dictionnaire étant une structure non ordonnée, Pandas ??????.
population = pd.Series([8.516, 67.06, 328.2, 1_386], index=["Suisse", "France", "USA", "Chine"])
area = pd.Series([41_285, 551_695, 3_796_742, 9_596_961], index=["Suisse", "France", "USA", "Chine"])
df1 = pd.DataFrame( { "Population" : population,
"Area" : area } )
df1| Population | Area | |
|---|---|---|
| Suisse | 8.516 | 41285 |
| France | 67.060 | 551695 |
| USA | 328.200 | 3796742 |
| Chine | 1386.000 | 9596961 |
dico = { "RS" : ["Facebook", "Twitter", "Instagram", "Linkedin", "Snapchat"],
"Budget" : [100, 50, 20, 100, 50],
"Audience": [1000, 300, 400, 50, 200],
"CPM" : [10, 20, 5, 200, 25] }
df1bis = pd.DataFrame(dico)
df1bis| RS | Budget | Audience | CPM | |
|---|---|---|---|---|
| 0 | 100 | 1000 | 10 | |
| 1 | 50 | 300 | 20 | |
| 2 | 20 | 400 | 5 | |
| 3 | 100 | 50 | 200 | |
| 4 | Snapchat | 50 | 200 | 25 |
1.3 Ligne par ligne à partir …
1.3.1 … d’une liste de Series [serie_1, serie_2, etc] ou d’une liste de dictionnaires [dico_1, dico_2, etc]
Chaque Series/Dictionnaire devient une ligne du DataFrame.
population = pd.Series(data=[8.516, 67.06, 328.2, 1_386], index=["Suisse", "France", "USA", "Chine"])
area = pd.Series(data=[41_285, 551_695, 3_796_742, 9_596_961], index=["Suisse", "France", "USA", "Chine"])
df1 = pd.DataFrame( data=[population, area],
index=["Population", "Area"])
df1| Suisse | France | USA | Chine | |
|---|---|---|---|---|
| Population | 8.516 | 67.06 | 328.2 | 1386.0 |
| Area | 41285.000 | 551695.00 | 3796742.0 | 9596961.0 |
population = {"Suisse":8.516, "France":67.06, "USA":328.2, "Chine":1_386}
area = {"Suisse":41_285, "France":551_695, "USA":3_796_742, "Chine":9_596_961}
df2 = pd.DataFrame( data=[population, area],
index=["Population", "Area"])
df2| Suisse | France | USA | Chine | |
|---|---|---|---|---|
| Population | 8.516 | 67.06 | 328.2 | 1386 |
| Area | 41285.000 | 551695.00 | 3796742.0 | 9596961 |
Si les deux dictionnaires ne contiennent pas les mêmes clés, les valeurs manquantes sont remplacées par NaN :
population = {"Suisse":8.516, "France":67.06, "USA":328.2}
area = {"Suisse":41_285, "France":551_695, "Chine":9_596_961}
df2bis = pd.DataFrame( data=[population, area],
index=["Population", "Area"])
df2bis| Suisse | France | USA | Chine | |
|---|---|---|---|---|
| Population | 8.516 | 67.06 | 328.2 | NaN |
| Area | 41285.000 | 551695.00 | NaN | 9596961.0 |
display(df2bis.values) # tableau numpy à 2 dimensions
display(df2bis.index) # objet de type Index
display(df2bis.columns) # objet de type Index
display(df2bis["France"]) # DataFrame["colonne"] renvoie une Series !!!! NB
display(df2bis["France"]["Population"]) # DataFrame["colonne"]["ligne"] !!! NB l'ordrearray([[8.516000e+00, 6.706000e+01, 3.282000e+02, nan],
[4.128500e+04, 5.516950e+05, nan, 9.596961e+06]])Index(['Population', 'Area'], dtype='object')Index(['Suisse', 'France', 'USA', 'Chine'], dtype='object')Population 67.06
Area 551695.00
Name: France, dtype: float6467.062 Accés aux éléments
| Population | Area | |
|---|---|---|
| Suisse | 8.516 | 41285 |
| France | 67.060 | 551695 |
| USA | 328.200 | 3796742 |
| Chine | 1386.000 | 9596961 |
display(df1.values) # tableau numpy à 2 dimensions
display(df1.index) # objet de type Index
display(df1.columns) # objet de type Indexarray([[8.516000e+00, 4.128500e+04],
[6.706000e+01, 5.516950e+05],
[3.282000e+02, 3.796742e+06],
[1.386000e+03, 9.596961e+06]])Index(['Suisse', 'France', 'USA', 'Chine'], dtype='object')Index(['Population', 'Area'], dtype='object')2.1 Selectionner une colonne : df['nom_colonne'] ou df.nom_colonne
Pour séléctionner la colonne col1 d’un DataFrame on peut utiliser df.col1, mais on préfèrera généralement df["col1"]. En effet, la première syntaxe ne fonctionne pas si le nom de la colonne contient des espaces ou des caractères spéciaux, ou si le nom de la colonne est identique à une méthode ou un attribut du DataFrame.
Le résultat est une Series.
# La colonne "Population" : c'est une Series
display(df1["Population"])
# Les colonnes "Population" et "Area" : c'est un DataFrame
display(df1[["Population", "Area"]])Suisse 8.516
France 67.060
USA 328.200
Chine 1386.000
Name: Population, dtype: float64| Population | Area | |
|---|---|---|
| Suisse | 8.516 | 41285 |
| France | 67.060 | 551695 |
| USA | 328.200 | 3796742 |
| Chine | 1386.000 | 9596961 |
Attention :
- dans un tableau numpy à 2 dimensions,
A[0]renvoi la première ligne du tableauA - pour un objet DataFrame, l’écriture
A[0]renvoi la première colonne.
C’est pourquoi il vaut mieux considérer un objet Dataframe comme un dictionnaire dont les clés sont les labels des colonnes plutôt que comme un tableau NumPy.
Une fois une colonne selectionnée (c’est une série), on peut selecionner un élément :
# La valeur de la cellule "Population" de la ligne "France"
col1 = "Population"
row1 = "France"
num_row1 = 1
display( df1[col1][row1] )
# display( df1[col1][num_row1] ) # deprecated
display( df1[col1].loc[row1] )
display( df1[col1].iloc[num_row1] )67.0667.0667.06Puisque les colonnes sont des Series, on peut utiliser les méthodes de Series sur les colonnes d’un DataFrame. En particulier, tout ce que l’on a dit sur le slicing des Series est valable pour les colonnes d’un DataFrame.
# SLICING
# =========
# Slicing sur les lignes: les valeurs de la colonne "Population" des lignes de "France" à "USA" inclus
display(df1["Population"]["France":"USA"])
# La colonne "Population", toutes les lignes
display(df1["Population"][:]) # idem que df1["Population"]
# Les valeurs de la ligne France
# ERROR : on ne peut pas extraire une ligne avec [:] !!!
# display(df1[:]["France"]) France 67.06
USA 328.20
Name: Population, dtype: float64Suisse 8.516
France 67.060
USA 328.200
Chine 1386.000
Name: Population, dtype: float642.2 Selectionner une ligne : loc et iloc
Si on veut selectionner une ligne, on utilise
- soit la méthode
.loc[label]qui utilise les labels et est la méthode recommandée ; - soit la méthode
.iloc[indice]qui utilise les indices de position (de \(0\) à \(N\) où \(N\) est égal àdf.shape[0]) et qui est deconsillée car les indices peuvent changer si on modifie le DataFrame.
Dans les deux cas, on obtient une Series dont les index sont les noms des colonnes. Ensuite, on pourra utiliser les méthodes de Series pour accéder aux éléments de la ligne.
NB Avec loc ou iloc, on peut accéder à un élément d’un DataFrame avec la syntaxe df.loc[label_ligne, nom_colonne] ou df.iloc[indice_ligne, indice_colonne] et retrouver ainsi l’ordre d’indexation des lignes et des colonnes de numpy : .iloc[i,j] renvoi l’élément de la ligne i et de la colonne j.
| Population | Area | |
|---|---|---|
| Suisse | 8.516 | 41285 |
| France | 67.060 | 551695 |
| USA | 328.200 | 3796742 |
| Chine | 1386.000 | 9596961 |
# Selectionner avec .loc (label de ligne)
# ========================================================
# Une ligne
display(df1.loc["France"]) # la ligne "France"
# Une cellule
display(df1.loc["France", "Population"]) # la valeur de la cellule "Population" de la ligne "France"
# Un slicing de lignes, une colonne
display(df1.loc["France":"USA", "Population"]) # les valeurs de la colonne "Population" des lignes de "France" à "USA" inclus
# Un slicing de lignes, plusieurs colonnes
display(df1.loc["France":"USA", ["Population", "Area"]]) # les valeurs des colonnes "Population" et "Area" des lignes de "France" à "USA" inclus
# Toutes les valeurs d'une colonne
display(df1.loc[:, "Population"]) # toutes les valeurs de la colonne "Population", idem que df1["Population"]
display(df1.iloc[:, 0]) # toutes les valeurs de la colonne "Population", idem que df1["Population"]Population 67.06
Area 551695.00
Name: France, dtype: float6467.06France 67.06
USA 328.20
Name: Population, dtype: float64| Population | Area | |
|---|---|---|
| France | 67.06 | 551695 |
| USA | 328.20 | 3796742 |
Suisse 8.516
France 67.060
USA 328.200
Chine 1386.000
Name: Population, dtype: float64Suisse 8.516
France 67.060
USA 328.200
Chine 1386.000
Name: Population, dtype: float64# Selectionner avec .iloc (indices de position)
# ========================================================
# Une ligne
display(df1.iloc[1]) # la ligne d'indice 1, toutes les colonnes
display(df1.iloc[1,:]) # la ligne d'indice 1, toutes les colonnes
# Une cellule
display(df1.iloc[1, 0]) # la valeur de la cellule d'indice 1, 0
# Un slicing de lignes, une colonne
display(df1.iloc[1:3, 0]) # les valeurs de la colonne "Population" des lignes d'indice 1 à 3 exclus
# Un slicing de lignes, plusieurs colonnes
display(df1.iloc[1:3, [0, 1]]) # les valeurs des colonnes "Population" et "Area" des lignes d'indice 1 à 3 exclus
# Toutes les valeurs d'une colonne
display(df1.iloc[:, 0]) # toutes les valeurs de la colonne "Population" idem que df1["Population"]Population 67.06
Area 551695.00
Name: France, dtype: float64Population 67.06
Area 551695.00
Name: France, dtype: float6467.06France 67.06
USA 328.20
Name: Population, dtype: float64| Population | Area | |
|---|---|---|
| France | 67.06 | 551695 |
| USA | 328.20 | 3796742 |
Suisse 8.516
France 67.060
USA 328.200
Chine 1386.000
Name: Population, dtype: float64On peut alors utiliser les masques de numpy etc.
display(df1)
# les lignes dont la Population est supérieure à 100, les colonnes "Population" et "Area"
# df1.loc[ df1["Population"] > 100, ["Population", "Area"] ]
mask = df1["Population"] > 100
display(mask) # une Series de booléens
df1.loc[ mask, : ] | Population | Area | |
|---|---|---|
| Suisse | 8.516 | 41285 |
| France | 67.060 | 551695 |
| USA | 328.200 | 3796742 |
| Chine | 1386.000 | 9596961 |
Suisse False
France False
USA True
Chine True
Name: Population, dtype: bool| Population | Area | |
|---|---|---|
| USA | 328.2 | 3796742 |
| Chine | 1386.0 | 9596961 |
2.2.1 Tableau comparatif entre numpy.ndarray et pandas.DataFrame
| Aspect | numpy.ndarray |
pandas.DataFrame |
|---|---|---|
| Nature | Tableau N-D homogène | Tableau 2D étiqueté (lignes + colonnes) |
| Types des données | ✔️ Un seul type par array | ✔️ Un type par colonne |
| Ordonnancement | ✔️ Ordonné | ✔️ Ordonné (index + colonnes) |
| Labels | ❌ Aucun | ✔️ Noms de colonnes + index |
| Accès positionnel | ✔️ arr[i, j] |
✔️ df.iloc[i, j] |
| Accès par label | ❌ | ✔️ df.loc[row_label, col_label] ou df.at[row_label, col_label] |
| Accès par colonne | ❌ | ✔️ df["nom_colonne"] (retourne une Series) |
| Accès par ligne | ❌ | ✔️ df.iloc[i] (par position), df.loc[row_label] (par label) |
| Slicing lignes | ✔️ arr[1:4] |
✔️ df.iloc[1:4] ou df.loc["y":"z"] |
| Slicing colonnes | ✔️ arr[:, 1:3] |
✔️ df[["col1","col2"]] ou df.loc[:, "col1":"col3"] |
| Vectorisation | ✔️ Très rapide | ✔️ Repose sur NumPy |
2.2.2 Résumé des principales syntaxes d’accès aux éléments d’un DataFrame :
| Syntaxe | Description |
|---|---|
df[eti_col] |
Sélectionne une colonne ou une séquence de colonnes. Peut aussi servir pour filtres booléens ou slices. |
df.loc[eti_ligne] |
Sélectionne une ou plusieurs lignes par leur étiquette de ligne (eti_ligne). |
df.loc[:, eti_col] |
Sélectionne une ou plusieurs colonnes par leurs étiquettes de colonnes (eti_col). |
df.loc[eti_ligne, eti_col] |
Sélectionne lignes (eti_ligne) et colonnes (eti_col) par leurs étiquettes. |
df.iloc[pos] |
Sélectionne une ou plusieurs lignes par leur position entière (pos). |
df.iloc[:, pos] |
Sélectionne une ou plusieurs colonnes par leur position entière (pos). |
df.iloc[pos_i, pos_j] |
Sélectionne lignes (pos_i) et colonnes (pos_j) par leurs positions entières. |
df.at[eti_ligne, eti_col] |
Accède à une valeur scalaire unique par étiquette de ligne (eti_ligne) et de colonne (eti_col). |
df.iat[i, j] |
Accède à une valeur scalaire unique par position entière de ligne (i) et de colonne (j). |
Méthode reindex |
Réorganise ou sélectionne lignes/colonnes par leurs étiquettes (eti_ligne et/ou eti_col). |
Méthodes get_value / set_value |
Accède ou modifie une valeur unique par étiquette (obsolètes, utilisez at/iat). |
2.3 Ajout de lignes/colonnes
2.3.1 Ajout d’une colonne
On ajoute une colonne à un DataFrame en utilisant la notation df['nom_colonne'] = serie, eventuellement en utilisant une colonne existante pour calculer la nouvelle colonne.
| Population | Area | |
|---|---|---|
| Suisse | 8.516 | 41285 |
| France | 67.060 | 551695 |
| USA | 328.200 | 3796742 |
| Chine | 1386.000 | 9596961 |
| Population | Area | Density | |
|---|---|---|---|
| Suisse | 8.516 | 41285 | 0.000206 |
| France | 67.060 | 551695 | 0.000122 |
| USA | 328.200 | 3796742 | 0.000086 |
| Chine | 1386.000 | 9596961 | 0.000144 |
Les colonnes sont toujours ajoutées à la fin du DataFrame. Si on veut spécifier une position (et décaler les autres colonnes) on utilise la méthode .insert() :
# ajout d'une colonne à une position donnée
df1.insert( 0, "DensityBIS", df1["Population"] / df1["Area"] )
df1| DensityBIS | Population | Area | Density | |
|---|---|---|---|---|
| Suisse | 0.000206 | 8.516 | 41285 | 0.000206 |
| France | 0.000122 | 67.060 | 551695 | 0.000122 |
| USA | 0.000086 | 328.200 | 3796742 | 0.000086 |
| Chine | 0.000144 | 1386.000 | 9596961 | 0.000144 |
2.3.2 Ajout d’une ligne
| Population | Area | |
|---|---|---|
| Suisse | 8.516 | 41285 |
| France | 67.060 | 551695 |
| USA | 328.200 | 3796742 |
| Chine | 1386.000 | 9596961 |
Pour ajouter une ligne à un DataFrame, on utilise la méthode .loc[label] = serie/liste/dict :
2.4 Réordonner les observations
La méthode sort_values permet de réordonner les observations d’un DataFrame, en laissant l’ordre des colonnes identiques. On peut trier selon une ou plusieurs colonnes, dans l’ordre croissant ou décroissant.
| Population | Area | |
|---|---|---|
| Suisse | 8.516 | 41285.0 |
| Italie | 60.360 | 301338.0 |
| France | 67.060 | 551695.0 |
| USA | 328.200 | 3796742.0 |
| Chine | 1386.000 | 9596961.0 |
2.5 Suppression de lignes/colonnes
Pour supprimer une ligne ou une colonne on utilise la méthode .drop() :
df.drop( nom_ligne, axis=0)oudf.drop(labels = nom_ligne)pour supprimer une lignedf.drop( nom_colonne, axis=1)oudf.drop( columns = nom_colonne)pour supprimer une colonne
On ajoute l’option inplace=True pour modifier le DataFrame, inplace=False pour renvoyer un nouveau DataFrame sans modifier l’original.
Attention, si la la ligne ou la colonne n’existe pas, une erreur sera levée. Pour éviter cela, on peut utiliser l’option errors='ignore'.
# Création du DataFrame
df1 = pd.DataFrame( { "Population" : population,
"Area" : area } )
df1.loc["Italie"] = [ 60.36, 301338]
df1["Density"] = df1["Population"] / df1["Area"]
df1.insert( 0, "DensityBIS", df1["Population"] / df1["Area"] )
df1| DensityBIS | Population | Area | Density | |
|---|---|---|---|---|
| Suisse | 0.000206 | 8.516 | 41285.0 | 0.000206 |
| France | 0.000122 | 67.060 | 551695.0 | 0.000122 |
| USA | 0.000086 | 328.200 | 3796742.0 | 0.000086 |
| Chine | 0.000144 | 1386.000 | 9596961.0 | 0.000144 |
| Italie | 0.000200 | 60.360 | 301338.0 | 0.000200 |
# Supprimoons une colonne
df1.drop( columns="DensityBIS", inplace=True ) # suppression d'une colonne
# del df1["Density"] # autre méthode pour supprimer une colonne
df1| Population | Area | Density | |
|---|---|---|---|
| Suisse | 8.516 | 41285.0 | 0.000206 |
| France | 67.060 | 551695.0 | 0.000122 |
| USA | 328.200 | 3796742.0 | 0.000086 |
| Chine | 1386.000 | 9596961.0 | 0.000144 |
| Italie | 60.360 | 301338.0 | 0.000200 |
3 Aperçu des données
Créons d’abord un DataFrame un peu plus richement rempli (on verra plus tard comment importer des données depuis un fichier CSV) :
data = pd.DataFrame( { "foo" : ["one", "one", "one", "two", "two", "two"],
"bar" : ["A", "A", "B", "A", "B", "B"],
"baz" : [1, 2, 3, 4, 5, 6],
"zoo" : ["x", "y", "z", "q", "w", 't'] } )
data| foo | bar | baz | zoo | |
|---|---|---|---|---|
| 0 | one | A | 1 | x |
| 1 | one | A | 2 | y |
| 2 | one | B | 3 | z |
| 3 | two | A | 4 | q |
| 4 | two | B | 5 | w |
| 5 | two | B | 6 | t |
3.1 Afficher les premières/dernières lignes
Un bon réflexe à adopter après la création/l’importation d’un DataFrale, et après toute transformation importante, est de visualiser le jeu de données, ou du moins quelques lignes, afin de vérifier que tout s’est correctement déroulé. Pour cela, il existe deux méthodes principales :
- la méthode
.head()permettant de sélectionner par défaut les 5 premières lignes du data frame. Il est possible de préciser entre parenthèses le nombre de lignes à afficher ; - la méthode
.tail()permettant de sélectionner par défaut les 5 dernières lignes du data frame. Il est également possible de préciser entre parenthèses le nombre de lignes à afficher.
Il n’est pas possible (par défaut) d’afficher plus de 60 lignes d’un data frame, afin de ne pas surcharger inutilement le notebook. De façon plus globale, chercher à visualiser l’ensemble d’un data frame n’est généralement pas une bonne pratique. Si cela est tout à fait envisageable avec quelques dizaines de lignes, ça devient vite impossible avec plusieurs millions de lignes ! Si vous cherchez à afficher plus de 60 lignes, vous aurez finalement comme résultat les 5 premières et dernières lignes du data frame.
| foo | bar | baz | zoo | |
|---|---|---|---|---|
| 0 | one | A | 1 | x |
| 1 | one | A | 2 | y |
| 2 | one | B | 3 | z |
| 3 | two | A | 4 | q |
| 4 | two | B | 5 | w |
3.2 Attributs d’un DataFrame
Combien de lignes comporte le DataFrame ? Et combien de colonnes ? Tout comme avec les arrays NumPy, il est possible de répondre à ces questions via l’attribut .shape. Le résultat est un tuple : le premier élément correspond au nombre de lignes, et le second au nombre de colonnes. On peut naturellement stocker le résultat de cet attribut dans une variable pour réutiliser ces éléments ultérieurement.
display(Markdown("**`ndim` : nombre de dimensions (2 pour un DataFrame)**"))
display( data.ndim)
display(Markdown("**`shape` : (nombre de lignes, nombre de colonnes)**"))
display( data.shape )
display(Markdown("**`size` : nombre total d'éléments (lignes * colonnes)**"))
display( data.size )
display(Markdown("**`axes` : liste des axes (index des lignes, index des colonnes)**"))
display( data.axes)
display(Markdown("**`index`, `columns`, `values` : accès aux composants du DataFrame**"))
display( data.index )
display( data.columns )
display( data.values ) # tableau numpy 2D des valeursndim : nombre de dimensions (2 pour un DataFrame)
2shape : (nombre de lignes, nombre de colonnes)
(6, 4)size : nombre total d’éléments (lignes * colonnes)
24axes : liste des axes (index des lignes, index des colonnes)
[RangeIndex(start=0, stop=6, step=1),
Index(['foo', 'bar', 'baz', 'zoo'], dtype='object')]index, columns, values : accès aux composants du DataFrame
RangeIndex(start=0, stop=6, step=1)Index(['foo', 'bar', 'baz', 'zoo'], dtype='object')array([['one', 'A', 1, 'x'],
['one', 'A', 2, 'y'],
['one', 'B', 3, 'z'],
['two', 'A', 4, 'q'],
['two', 'B', 5, 'w'],
['two', 'B', 6, 't']], dtype=object)On peut avoir envie de connaître les types de chacune de nos variables (un type par colonne). On peut accéder à cela très simplement à partir de l’attribut .dtypes. On obtient un objet Series :les index de cette série sont les étiquettes des colonnes, et les valeurs de cette série sont les types de données. Vous noterez que le type de foo est objet, alors que nous avons pourtant des chaînes de caractères. C’est une chose à connaître, mais le type objet de Pandas correspond en fait à une colonne de type chaîne de caractères.
Les jeux de données réels sont rarement complets et les valeurs manquantes peuvent refléter de nombreuses réalités : problème de remontée d’information, variable non pertinente pour cette observation, etc.
Par défaut, les valeurs manquantes sont affichées NaN et sont de type np.nan. On a un comportement cohérent d’agrégation lorsqu’on combine deux colonnes dont l’une comporte des valeurs manquantes.
Pour connaitre le nombre de valeurs manquantes dans chaque colonne, on peut utiliser la méthode .isnull().
Il est possible de supprimer les valeurs manquantes grâce à .dropna(). Cette méthode va supprimer toutes les lignes où il y a au moins une valeur manquante.
On peut sinon donner une valeur aux valeurs manquantes grâce à la méthode .fillna(), par exemple en prénant la moyenne de la colonne.
| foo | bar | baz | zoo | |
|---|---|---|---|---|
| 0 | False | False | False | False |
| 1 | False | False | False | False |
| 2 | False | False | False | False |
| 3 | False | False | False | False |
| 4 | False | False | False | False |
| 5 | False | False | False | False |
Ces informations peuvent être combinées pour obtenir un résumé complet des données avec la méthode .info().
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 6 entries, 0 to 5
Data columns (total 4 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 foo 6 non-null object
1 bar 6 non-null object
2 baz 6 non-null int64
3 zoo 6 non-null object
dtypes: int64(1), object(3)
memory usage: 324.0+ bytes3.3 Statistiques descriptives
On peut calculer la somme, la plus petite/grande valeur, la moyenne, la médiane, l’écart-type des élements de chaque colonne avec les méthodes .sum(), .min(), .max(), .mean(), .median() et .std().
Rappel : la somme entre chaînes de caractères est une concaténation.
# Elles renvoient des Series dont les Index sont les colonnes du DataFrame
display(Markdown("**Opérations sur les DataFrames**"))
display(Markdown("**Somme**"))
display(data.sum()) # idem que data.sum(axis=0) ou data.sum(axis='rows')
display(Markdown("**Minimum**"))
display(data.min())
display(Markdown("**Maximum**"))
display(data.max())
display(Markdown("**Moyenne**"))
# display(data.mean())
display(data.mean(numeric_only=True))
display(Markdown("**Médiane**"))
# display(data.median())
display(data.median(numeric_only=True))
display(Markdown("**Écart-type**"))
# display(data.std())
display(data.std(numeric_only=True))
display(Markdown("**Comptage : nombre de valeurs non manquants par colonne**"))
display(data.count())Opérations sur les DataFrames
Somme
foo oneoneonetwotwotwo
bar AABABB
baz 21
zoo xyzqwt
dtype: objectMinimum
foo one
bar A
baz 1
zoo q
dtype: objectMaximum
foo two
bar B
baz 6
zoo z
dtype: objectMoyenne
baz 3.5
dtype: float64Médiane
baz 3.5
dtype: float64Écart-type
baz 1.870829
dtype: float64Comptage : nombre de valeurs non manquants par colonne
foo 6
bar 6
baz 6
zoo 6
dtype: int64Pour obtenir un résumé statistique des variables numériques, on peut utiliser la méthode .describe() :
| baz | |
|---|---|
| count | 6.000000 |
| mean | 3.500000 |
| std | 1.870829 |
| min | 1.000000 |
| 25% | 2.250000 |
| 50% | 3.500000 |
| 75% | 4.750000 |
| max | 6.000000 |
Cette méthode renvoie un objet de type DataFrame, où les statistiques descriptives sont calculées pour chaque colonne numérique. Pour les variables catégorielles, on peut obtenir le nombre de valeurs uniques, la valeur la plus fréquente et sa fréquence :
| foo | bar | baz | zoo | |
|---|---|---|---|---|
| count | 6 | 6 | 6.000000 | 6 |
| unique | 2 | 2 | NaN | 6 |
| top | one | A | NaN | x |
| freq | 3 | 3 | NaN | 1 |
| mean | NaN | NaN | 3.500000 | NaN |
| std | NaN | NaN | 1.870829 | NaN |
| min | NaN | NaN | 1.000000 | NaN |
| 25% | NaN | NaN | 2.250000 | NaN |
| 50% | NaN | NaN | 3.500000 | NaN |
| 75% | NaN | NaN | 4.750000 | NaN |
| max | NaN | NaN | 6.000000 | NaN |
On peut modifier le type de données d’une colonne avec la méthode .astype() et demander à Pandas d’y associer des codes numériques. Si on ne veut pas ecraser le DataFrame initial, on peut utiliser la méthode .copy().
data_bis = data.copy()
for col in data_bis.select_dtypes(include='object').columns:
data_bis[col] = data_bis[col].astype('category').cat.codes
display(data)
display(data_bis)| foo | bar | baz | zoo | |
|---|---|---|---|---|
| 0 | one | A | 1 | x |
| 1 | one | A | 2 | y |
| 2 | one | B | 3 | z |
| 3 | two | A | 4 | q |
| 4 | two | B | 5 | w |
| 5 | two | B | 6 | t |
| foo | bar | baz | zoo | |
|---|---|---|---|---|
| 0 | 0 | 0 | 1 | 3 |
| 1 | 0 | 0 | 2 | 4 |
| 2 | 0 | 1 | 3 | 5 |
| 3 | 1 | 0 | 4 | 0 |
| 4 | 1 | 1 | 5 | 2 |
| 5 | 1 | 1 | 6 | 1 |
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 6 entries, 0 to 5
Data columns (total 4 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 foo 6 non-null int8
1 bar 6 non-null int8
2 baz 6 non-null int64
3 zoo 6 non-null int8
dtypes: int64(1), int8(3)
memory usage: 198.0 bytes| foo | bar | baz | zoo | |
|---|---|---|---|---|
| count | 6.000000 | 6.000000 | 6.000000 | 6.000000 |
| mean | 0.500000 | 0.500000 | 3.500000 | 2.500000 |
| std | 0.547723 | 0.547723 | 1.870829 | 1.870829 |
| min | 0.000000 | 0.000000 | 1.000000 | 0.000000 |
| 25% | 0.000000 | 0.000000 | 2.250000 | 1.250000 |
| 50% | 0.500000 | 0.500000 | 3.500000 | 2.500000 |
| 75% | 1.000000 | 1.000000 | 4.750000 | 3.750000 |
| max | 1.000000 | 1.000000 | 6.000000 | 5.000000 |
3.4 Comptage : valeurs uniques, dénombrement de valeurs et appartenance
Une autre catégorie de méthodes connexes permet d’extraire des informations sur les valeurs contenues dans une série unidimensionnelle. Il s’agit des méthodes .count(), .unique(), .nunique() et .value_counts().
# Combien de valuers : ici , on peut également utiliser .size(), puisqu'il n'y a pas de valeurs manquantes
data['foo'].count()6Pour déterminer le nombre de valeurs uniques d’une variable, plutôt que chercher à écrire soi-même une fonction, on utilise la méthode nunique.
foo 2
bar 2
baz 6
zoo 6
dtype: int64# Pour chaque colonne selectionnée, on affiche les valeurs uniques :
for col in data.columns:
print((f"\nValeurs uniques dans la colonne {col} :"))
print(data[col].unique())
Valeurs uniques dans la colonne foo :
['one' 'two']
Valeurs uniques dans la colonne bar :
['A' 'B']
Valeurs uniques dans la colonne baz :
[1 2 3 4 5 6]
Valeurs uniques dans la colonne zoo :
['x' 'y' 'z' 'q' 'w' 't']# On peut obtenir ces deux informations en une seule instruction :
for col in data.columns:
print((f"\nValeurs et occurrences dans la colonne {col} :"))
print(data[col].value_counts())
Valeurs et occurrences dans la colonne foo :
foo
one 3
two 3
Name: count, dtype: int64
Valeurs et occurrences dans la colonne bar :
bar
A 3
B 3
Name: count, dtype: int64
Valeurs et occurrences dans la colonne baz :
baz
1 1
2 1
3 1
4 1
5 1
6 1
Name: count, dtype: int64
Valeurs et occurrences dans la colonne zoo :
zoo
x 1
y 1
z 1
q 1
w 1
t 1
Name: count, dtype: int644 Filtres et opérations
L’opération de sélection de lignes s’appelle Filtrer. Elle s’utilise en fonction d’une condition logique, on sélectionne les données sur une condition logique.
Il existe plusieurs méthodes en Pandas. La plus simple est d’utiliser les boolean mask, déjà vus en numpy.
La plupart de ce qu’on a pu voir avec les arrays de NumPy est applicable aux objets Series et DataFrames de Pandas. On peut par exemple effectuer des opérations arithmétiques sur les objets, ou encore appliquer des fonctions mathématiques.
4.1 Opérations sur les DataFrames
On peut appliquer des opérations arithmétiques sur les DataFrames, de la même manière que pour les Series. Les opérations sont effectuées élément par élément, en alignant les lignes et les colonnes par leurs étiquettes respectives.
| Population | Area | Density | NewCol1 | NewCol2 | |
|---|---|---|---|---|---|
| Suisse | 8.516 | 41285.0 | 0.000206 | 17.032 | 0.000206 |
| France | 67.060 | 551695.0 | 0.000122 | 134.120 | 0.000122 |
| USA | 328.200 | 3796742.0 | 0.000086 | 656.400 | 0.000086 |
| Italie | 60.360 | 301338.0 | 0.000200 | 120.720 | 0.000200 |
4.2 Filtrer un DataFrame : masque booléen vs query()
Pandas propose deux façons principales de filtrer un DataFrame : les masques booléens “à la NumPy”, très flexibles, et la méthode query(), plus lisible et proche du SQL.
Voici un exemple de DataFrame que nous allons utiliser pour illustrer ces deux approches :
4.2.1 Filtre “à la NumPy” (masques booléens)
Avec cette méthode, on crée un masque booléen en appliquant une condition logique sur une ou plusieurs colonnes du DataFrame. On utilise ensuite ce masque pour sélectionner les lignes correspondantes.
- Avantages
- Très flexible (méthodes pandas/NumPy, fonctions Python).
- Compatible avec tous les noms de colonnes.
- Inconvénients
- Syntaxe moins intuitive car
& | ~au lieu deand or notet parenthèses obligatoires.
- Syntaxe moins intuitive car
# `&` pour le `and`
# `|` pour le `or`
# `!` pour le `not`
# parenthèses obligatoires
mask = (data["bar"]=="A") & (data["baz"]>=2)
data[mask]| foo | bar | baz | zoo | |
|---|---|---|---|---|
| 1 | one | A | 2 | y |
| 3 | two | A | 4 | q |
Remarque : dans l’exemple ci-dessus, on utilise la méthode str.startswith('t') pour vérifier si les chaînes de caractères de la colonne ‘foo’ commencent par la lettre ‘t’. .str. est une méthode particulière qui permet de traiter chaque valeur d’une colonne comme un string natif en Python sur lequel appliquer une méthode ultérieure (en l’occurrence startswith).
4.2.2 Filtre avec query()
- Avantages
- Syntaxe type SQL : plus lisible.
- Pas de parenthèses autour des conditions.
- Inconvénients
- Limitations si noms de colonnes avec espaces ou fonctions Python complexes.
- Expression passée en chaîne de caractères.
5 Aggregations
5.1 groupby : analyse par groupes
La méthode groupby permet de grouper les données suivant certains critères.
Commençons par un exemple simple :
df = pd.DataFrame({'key': ['A', 'B', 'C', 'A', 'B', 'C'],
'data': range(6)}, columns=['key', 'data'])
display(Markdown("**DataFrame initial**"))
display(df)
display(Markdown("**GroupBy sum sur la colonne 'key'**"))
display(df.groupby('key').sum())DataFrame initial
| key | data | |
|---|---|---|
| 0 | A | 0 |
| 1 | B | 1 |
| 2 | C | 2 |
| 3 | A | 3 |
| 4 | B | 4 |
| 5 | C | 5 |
GroupBy sum sur la colonne ‘key’
| data | |
|---|---|
| key | |
| A | 3 |
| B | 5 |
| C | 7 |
df = pd.DataFrame({'key': ['A', 'B', 'C', 'A', 'B', 'C'],
'data': range(6),
'toto': ['a','a','b','b','a','b']},
columns=['key', 'data', 'toto'])
display(Markdown("**DataFrame initial**"))
display(df)
display(Markdown("**GroupBy sum sur la colonne 'key'**"))
display(df.groupby('key').sum())DataFrame initial
| key | data | toto | |
|---|---|---|---|
| 0 | A | 0 | a |
| 1 | B | 1 | a |
| 2 | C | 2 | b |
| 3 | A | 3 | b |
| 4 | B | 4 | a |
| 5 | C | 5 | b |
GroupBy sum sur la colonne ‘key’
| data | toto | |
|---|---|---|
| key | ||
| A | 3 | ab |
| B | 5 | aa |
| C | 7 | bb |
C’est ce que l’on appelle le paradigme « split-apply-combine » (diviser-appliquer-combiner) : d’abord on génère un groupe de DataFrame en fonction de la valeur de la clé spécifiée. À chaque groupe, on applique ensuite une fonction d’agrégation (ici la somme) pour obtenir une seule ligne. Enfin on fusionne les lignes obtenues dans un nouveau DataFrame.
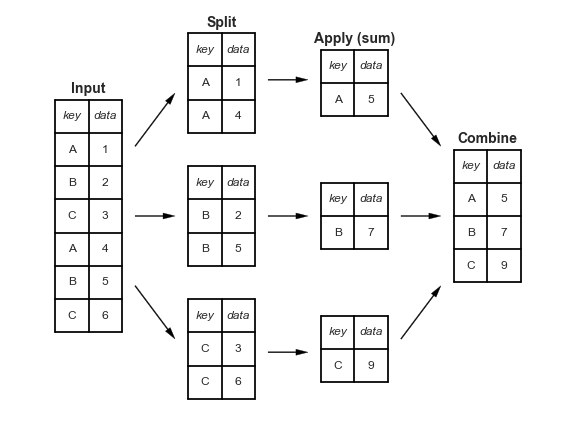
Passons à un exemple plus riche.
data = pd.DataFrame( { "foo" : ["one", "one", "one", "two", "two", "two"],
"bar" : ["A", "A", "B", "A", "B", "B"],
"baz" : [1, 2, 3, 4, 5, 6],
"zoo" : ["x", "y", "z", "q", "w", 't'] } )
data| foo | bar | baz | zoo | |
|---|---|---|---|---|
| 0 | one | A | 1 | x |
| 1 | one | A | 2 | y |
| 2 | one | B | 3 | z |
| 3 | two | A | 4 | q |
| 4 | two | B | 5 | w |
| 5 | two | B | 6 | t |
On va regrouper les données en fonction de la colonne foo.
<pandas.core.groupby.generic.DataFrameGroupBy object at 0x783e02c53050>On obtient un objet DataFrameGroupBy, c’est-à-dire une sorte de dictionnaire de DatFrame dont les clés sont les valeurs uniques de la colonne foo et les valeurs sont les DataFrame correspondant à chaque groupe :
Nom du groupe : one
| foo | bar | baz | zoo | |
|---|---|---|---|---|
| 0 | one | A | 1 | x |
| 1 | one | A | 2 | y |
| 2 | one | B | 3 | z |
Nom du groupe : two
| foo | bar | baz | zoo | |
|---|---|---|---|---|
| 3 | two | A | 4 | q |
| 4 | two | B | 5 | w |
| 5 | two | B | 6 | t |
On peut regrouper les données en fonction de plusieurs colonnes. Par exemple, on peut regrouper les données en fonction des colonnes foo et bar :
data_gbfoobar = data.groupby(['foo', 'bar'])
for name, group in data_gbfoobar:
display(Markdown(f"**Nom du groupe : {name}**"))
display(group)Nom du groupe : (‘one’, ‘A’)
| foo | bar | baz | zoo | |
|---|---|---|---|---|
| 0 | one | A | 1 | x |
| 1 | one | A | 2 | y |
Nom du groupe : (‘one’, ‘B’)
| foo | bar | baz | zoo | |
|---|---|---|---|---|
| 2 | one | B | 3 | z |
Nom du groupe : (‘two’, ‘A’)
| foo | bar | baz | zoo | |
|---|---|---|---|---|
| 3 | two | A | 4 | q |
Nom du groupe : (‘two’, ‘B’)
| foo | bar | baz | zoo | |
|---|---|---|---|---|
| 4 | two | B | 5 | w |
| 5 | two | B | 6 | t |
On peut appliquer une même opération sur chaque groupe en même temps. Par exemple, on peut extraire une seule colonne de cet objet, et on obtient un objet SeriesGroupBy :
<pandas.core.groupby.generic.SeriesGroupBy object at 0x783e018e6630>On obtient ainsi un objet SeriesGroupBy, qui est une sorte de dictionnaire de Series. Chaque Series correspond à un groupe.
Nom du groupe : one
0 1
1 2
2 3
Name: baz, dtype: int64Nom du groupe : two
3 4
4 5
5 6
Name: baz, dtype: int64Pour ne pas itérer sur les groupes mais juste connaître des informations sur les groupes, on peut utiliser :
ngroupspermet de connaître le nombre de groupes obtenus (ici 2 car on a deux valeurs uniques dans la colonnefoo)sizecombien d’éléments sont dans chaque groupegroupsles indices des lignes de chaque groupe avec la méthode.
# Combien de groupes ?
nb_groupe = data_gbfoo.ngroups
display(nb_groupe)
# Combien d'éléments par groupe ?
nb_element_par_groupe = data_gbfoo.size()
display(nb_element_par_groupe)
# Quelles sont les indices des lignes de chaque groupe ?
group_indices = data_gbfoo.groups
display(group_indices)2foo
one 3
two 3
dtype: int64{'one': [0, 1, 2], 'two': [3, 4, 5]}L’object DataFrameGroupBy peut être immaginé comme un dictionnaire de DataFrames où chaque clé est un nom de groupe et chaque valeur est le DataFrame correspondant à ce groupe. Cependant, ce c’est pas un vrai dictionnaire, en particulier on ne peut pas accéder aux groupes avec la syntaxe des dictionnaires data_gbfoo['one'].
Pour récuperer les dataframes on doit utiliser la méthode get_group() :
data_one = data_gbfoo.get_group("one") # les lignes du groupe "one" constitue un dataframe
data_two = data_gbfoo.get_group("two") # les lignes du groupe "two" constitue un dataframe
display(data_one, data_two)| foo | bar | baz | zoo | |
|---|---|---|---|---|
| 0 | one | A | 1 | x |
| 1 | one | A | 2 | y |
| 2 | one | B | 3 | z |
| foo | bar | baz | zoo | |
|---|---|---|---|---|
| 3 | two | A | 4 | q |
| 4 | two | B | 5 | w |
| 5 | two | B | 6 | t |
L’utilisation principale de groupby nest pas la simple création de sous-DataFrames. Généralement on applique des fonctions d’agrégation sur les groupes obtenus. On obtient un unique DataFrame avec une ligne par groupe.
Par exemple, on peut calculer la somme des éléments de chaque groupe (on se rappelle que l’operation “somme” entre chaînes de caractères correspond à la concaténation de ces chaînes) :
On peut aussi regrouper par plusieurs colonnes, puis appliquer une fonction d’agrégation.
Exemple : regroupons les données de data en utilisant les colonnes foo et bar, puis calculons la somme de la colonne baz pour chaque groupe unique de foo et bar.
Voici les étapes décomposées :
data_grfoobar = data.groupby(['foo', 'bar']): regroupe le DataFrame par les colonnesfooetbar, en obtenant autant de groupes que de couples différents ;data_grfoobar['baz'].sum(): calcule la somme debazpour chaque groupe.- Facultatif :
.unstack()qui transforme le résultat en une table pivotée, en déplaçant les valeurs uniques debaren colonnes. Ainsi, pour chaque valeur defoo, on obtient les sommes debazréparties par les différentes valeurs debar.
Le résultat est un DataFrame avec les valeurs de foo en index et celles de bar en colonnes, où chaque cellule contient la somme correspondante de baz.
# on regroupe les couples (foo, bar)
data_grfoobar = data.groupby(['foo', 'bar'])
display(data_grfoobar.get_group(('one', 'A'))) # df pour qui foo == 'one' et bar == 'A'
display(data_grfoobar.get_group(('one', 'B'))) # df pour qui foo == 'one' et bar == 'B'
display(data_grfoobar.get_group(('two', 'A'))) # df pour qui foo == 'two' et bar == 'A'
display(data_grfoobar.get_group(('two', 'B'))) # df pour qui foo == 'two' et bar == 'B'| foo | bar | baz | zoo | |
|---|---|---|---|---|
| 0 | one | A | 1 | x |
| 1 | one | A | 2 | y |
| foo | bar | baz | zoo | |
|---|---|---|---|---|
| 2 | one | B | 3 | z |
| foo | bar | baz | zoo | |
|---|---|---|---|---|
| 3 | two | A | 4 | q |
| foo | bar | baz | zoo | |
|---|---|---|---|---|
| 4 | two | B | 5 | w |
| 5 | two | B | 6 | t |
# Pour chaque groupe, on somme les valeurs de la colonne "baz"
data_gbfoobarbaz = data_grfoobar['baz'].sum()
data_gbfoobarbaz # c'est une Series avec un MultiIndexfoo bar
one A 3
B 3
two A 4
B 11
Name: baz, dtype: int64MultiIndex([('one', 'A'),
('one', 'B'),
('two', 'A'),
('two', 'B')],
names=['foo', 'bar'])Avec .unstack(), on peut transformer la Series MultiIndex en DataFrame pour une meilleure lisibilité :
5.2 pivot_table : tableaux croisés
Le résultat obtenu avec l’instruction :
data.groupby(['col1', 'col2'])['col3'].sum().unstack()
est un tableau croisé dynamique : à chaque couple (col1, col2), on associe la somme des valeurs de col3.
Si le DataFrame data contient les colonnes col1, col2 et col3, on peut imaginer que chaque ligne du DataFrame correspond à un triplet (x_i, y_i, z_i). On peut alors representer cela comme un point en 3D :
col1est la coordonnéex_icol2est la coordonnéey_icol3est la coordonnéez_i
On peut imaginer que l’on cherche à représenter une fonction de deux variables \(z = f(x,y)\), où \(x\) et \(y\) sont les coordonnées dans le plan horizontal, et \(z\) est la hauteur associée à chaque point \((x,y)\). Problème : si le couple (x_i, y_i) est unique dans le DataFrame, on a un seul point pour cette position. Mais si plusieurs lignes \(j\) ont le même couple (x_i, y_i) mais des valeurs différentes (z_i)_j, cela correspond à plusieurs point empilés verticalement, ce qui ne représente plus une fonction de \(\mathbb R^2\) vers \(\mathbb R\). Dans ce cas, pour obtenir une valeur unique \(\tilde z_i\), il faut agréger ces différentes valeurs. C’est là qu’intervient l’agrégation (ici la somme) :
\[ (x_i, y_i) \mapsto \tilde z_i = \sum_{j} (z_i)_j \]
On pourrait également utiliser d’autres fonctions d’agrégation comme mean, max, min, etc.
C’est une opération très courante en analyse de données. Pandas propose donc une méthode dédiée : pivot_table.
On applique la méthode pivot_table sur le DataFrame data en précisant :
index: le nom de la colonne dans de DataFrame qui sera la ligne du nouveau tableaucolumns: le nom de la colonne dans le DataFrame qui sera la colonne du nouveau tableauvalues: le nom de la colonne sur laquelle appliquer l’agrégationaggfunc: la fonction d’agrégation (sum,mean,count,min,max, etc.). Si ce n’est pas spécifié, par défaut on calcule la moyenne (mean) des valeurs.
Avec pivot_table, on obtient un résultat équivalent à
groupby(...).agg(...).unstack()
mais plus lisible et directement structuré sous forme de tableau croisé.
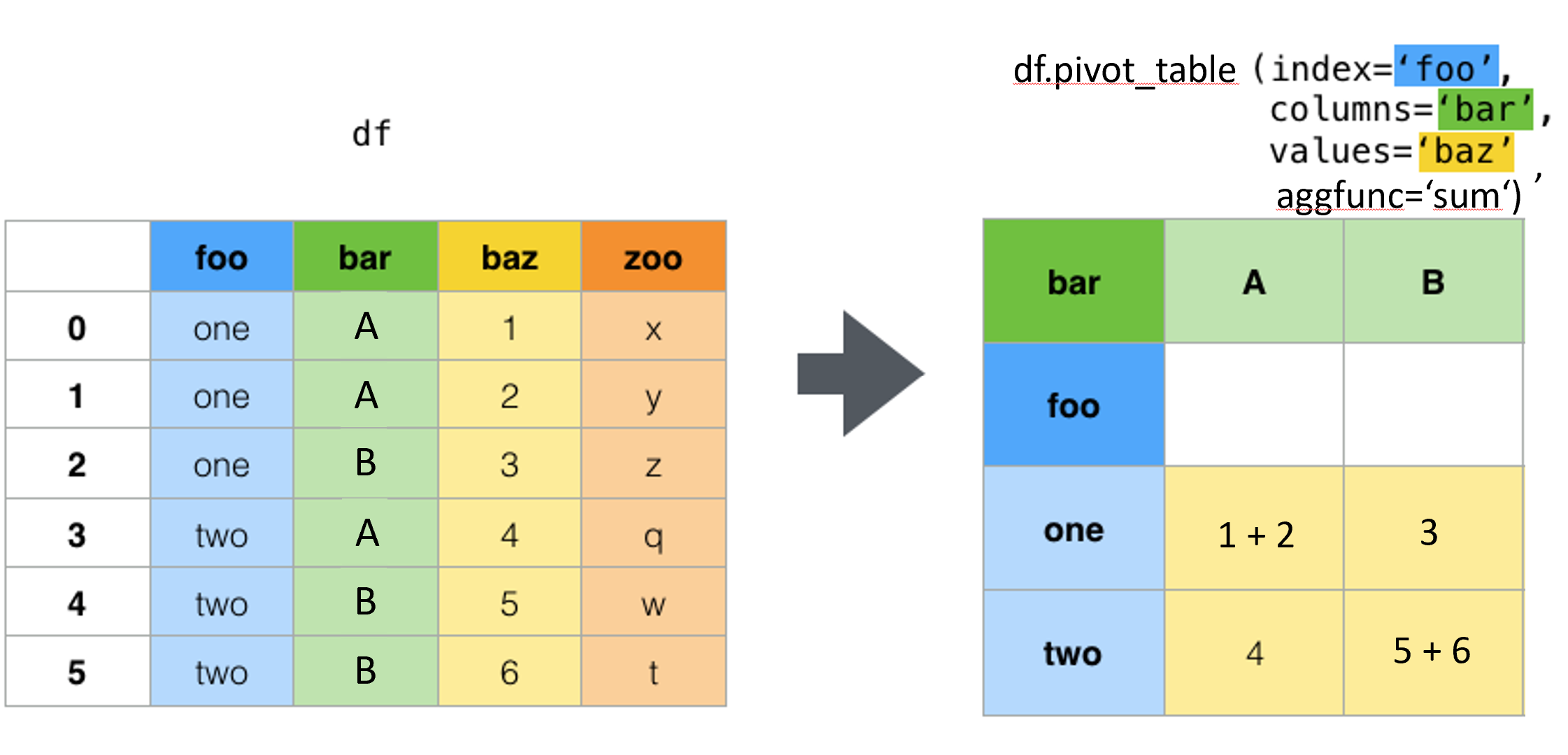
| bar | A | B |
|---|---|---|
| foo | ||
| one | 3 | 3 |
| two | 4 | 11 |
Lorsqu’on a besoin de calculer des sous-totaux, on peut utiliser l’option margins=True :
5.3 crosstab : tableaux croisés
crosstab est une fonction principalement utilisée pour compter les occurrences ou les fréquences des combinaisons de valeurs de deux colonnes.
Cette fonction crée un tableau croisé indipendamment d’un DataFrame, en utilisant deux séries ou colonnes comme entrées :
index: les données stockées dans une séries ou une colonne d’un DataFrame qui sera la ligne du nouveau tableaucolumns: les données stockées dans une séries ou une colonne d’un DataFrame qui sera la colonne du nouveau tableau
Pour chaque combinaison unique de valeurs dans col1 et col2, crosstab compte le nombre d’occurrences et remplit le tableau avec ces comptes.
crosstab peut également agréger les valeurs d’une colonne supplémentaire, mais la syntaxe est différente de pivot_table :
indexetcolumns: comme précédemmentvalues: les données stockées dans une séries ou une colonne d’un DataFrame qui sera la colonne à agrégeraggfunc: fonction d’agrégation (par défautcountpour compter les occurrences, mais on peut utilisersum,mean, etc.)
Enfin, crosstab propose un argument très pratique, normalize, qui permet d’afficher les résultats en pourcentages (par rapport à la somme totale, aux lignes ou aux colonnes) en une seule instruction, sans calcul supplémentaire.
La commande pd.crosstab(data['foo'], data['bar']) crée un tableau croisé (ou “contingence”) en comptant les occurrences de chaque combinaison unique de valeurs dans les colonnes foo et bar du DataFrame data.
pd.crosstab(data['foo'], data['bar']): compte le nombre de fois que chaque paire de valeurs (foo,bar) apparaît dans le DataFrame.- Le résultat est un nouveau DataFrame avec :
- Les valeurs uniques de
foocomme index (lignes). - Les valeurs uniques de
barcomme colonnes. - Les cellules contiennent le nombre d’occurrences de chaque combinaison
(foo, bar).
- Les valeurs uniques de
| bar | A | B |
|---|---|---|
| foo | ||
| one | 2 | 1 |
| two | 1 | 2 |
C’est utile pour obtenir une vue d’ensemble des fréquences d’association entre les deux variables. Pour obtenir les fréquences au lieu des comptes bruts dans un tableau croisé de pandas, on ajoute l’argument normalize=True, chaque valeur est alors divisée par le total de toutes les combinaisons. Cela affichera chaque valeur comme une proportion du total, ce qui correspond aux fréquences relatives.
| bar | A | B |
|---|---|---|
| foo | ||
| one | 0.333333 | 0.166667 |
| two | 0.166667 | 0.333333 |
On peut aussi normaliser par ligne ou par colonne :
- Par ligne (chaque ligne totalisera 1):
normalize='index' - Par colonne (chaque colonne totalisera 1):
normalize='columns'
5.4 Arithmétique et alignement des données : différences avec Numpy
Il existe une différence très importante : lorsque vous faites une opération entre deux objets Series, l’opération se fait terme à terme, mais les termes sont identifiés par leur index. Si les index ne correspondent pas, le résultat sera un objet Series avec un index qui est l’union des deux index initiaux. Les valeurs correspondant à des index qui n’étaient pas présents dans les deux objets initiaux seront des valeurs manquantes, notées NaN (pour Not a Number).
ma_serie4 = pd.Series( np.random.randn(5), index=["A","B","C","D","E"] )
display(ma_serie4)
ma_serie5 = pd.Series( np.random.randn(4), index=["A","B","C","F"] )
display(ma_serie5)
ma_serie_somme = ma_serie4 + ma_serie5
display(ma_serie_somme)A 0.696382
B 0.804378
C 0.834349
D 0.729562
E 0.273123
dtype: float64A 1.308273
B 0.598953
C -0.667426
F 1.263611
dtype: float64A 2.004655
B 1.403331
C 0.166923
D NaN
E NaN
F NaN
dtype: float64On voit ici que les deux Series ont en commun A, B et C. La somme donne bien une valeur qui est la somme des deux objets Series. Pour D, E et F, on obtient une valeur manquante car l’un des deux objets Series ne comprend pas d’index D, E ou F. On a donc la somme d’une valeur manquante et d’une valeur présente qui logiquement donne une valeur manquante. Si vous voulez faire une somme en supposant que les données manquantes sont équivalentes à 0, il faut utiliser : s1.add(s2, fill_value=0):
5.5 L’indexation des DataFrames
Vous pouvez avoir besoin de modifier les index de vos données. Pour cela, différentes options s’offrent à vous :
.reindex()permet de sélectionner des colonnes et de réordonner les colonnes et les lignes. Si une colonne ou une ligne n’est pas présente dans le DataFrame initial, elle sera ajoutée avec des valeurs manquantes.
df5 = pd.DataFrame( np.random.randn(5,2), index=["a","b","c","d","e"], columns=["Col 1","Col 2"])
df5| Col 1 | Col 2 | |
|---|---|---|
| a | -0.844568 | 1.395210 |
| b | -0.171219 | 3.200248 |
| c | 1.836659 | -0.828000 |
| d | 0.394072 | 1.467102 |
| e | -1.848099 | 0.137949 |
# on ne garde que les lignes "c","d","e" mais on les réordonne
# on garde toutes les colonnes mais on les réordonne aussi
df6 = df5.reindex(index=["e","c","d"], columns=["Col 2","Col 1"])
df6| Col 2 | Col 1 | |
|---|---|---|
| e | 0.137949 | -1.848099 |
| c | -0.828000 | 1.836659 |
| d | 1.467102 | 0.394072 |
.rename()permet de renommer les colonnes ou les lignes.
| Seconde | Premiere | |
|---|---|---|
| E | 0.137949 | -1.848099 |
| C | -0.828000 | 1.836659 |
| d | 1.467102 | 0.394072 |
| Seconde | Premiere | |
|---|---|---|
| E | 0.137949 | -1.848099 |
| C | -0.828000 | 1.836659 |
| D | 1.467102 | 0.394072 |
5.6 Copie VS vue
Au même titre que les objets de NumPy (ou les listes tout simplement), il est important de comprendre comment les objets Series et DataFrame sont alloués. Lorsqu’on crée un objet DataFrame ou Series à partir d’un autre objet, le fait de savoir si on a affaire à une copie ou à une référence dépend de l’objet d’origine. Lorsqu’on travaille sur un array, il s’agit juste d’une référence aux valeurs.
Dans l’exemple suivant, on voit que l’array initial est impacté par la modification de l’objet DataFrame. Si vous faites la même chose avec une liste, Pandas crée une copie.
Une fois que vous avez créé votre DataFrame, si vous allouez le même DataFrame à un objet, il va faire référence au premier DataFrame.
Si vous créez un DataFrame à partir d’une partie de votre DataFrame, vous obtiendrez une vue de votre DataFrame.
Conclusion, si vous voulez réellement une copie, il faudra utiliser la méthode .copy() mais soyez attentifs à l’espace nécessaire. Vous pouvez créer des vues comme avec NumPy en utilisant .view().
# on crée un array
arr1 = np.arange(6).reshape(3,2)
# on crée un DataFrame à partir de l’array
df1 = pd.DataFrame(arr1)
print("Avant modification")
display(arr1)
display(df1)
print("Après modification d'une valeur du DataFrame")
df1.iloc[1,1] = 22
display(df1)
display(arr1) # <--- l'array aussi A ÉTÉ MODIFIÉAvant modification
Après modification d'une valeur du DataFramearray([[0, 1],
[2, 3],
[4, 5]])| 0 | 1 | |
|---|---|---|
| 0 | 0 | 1 |
| 1 | 2 | 3 |
| 2 | 4 | 5 |
| 0 | 1 | |
|---|---|---|
| 0 | 0 | 1 |
| 1 | 2 | 22 |
| 2 | 4 | 5 |
array([[ 0, 1],
[ 2, 22],
[ 4, 5]])